DNA-Isolierung und Plasmid-Isolierung
DNA-Isolierung /
Plasmid-Isolierung /
Videos
Hinweis:
Im Atelier finden Sie die CD "Gentechnik, interaktiv
experimentieren". Sie betreten (vituell) das Biozentrum Basel und
führen dort ein Klonierungsexperiment durch.
DNA kann leicht aus eukaryotischen und prokaryotischen Zellen
isoliert werden. Die Isolierung von DNA stellt den ersten Schritt im
Arbeitsprozess einer Gen-Klonierung dar. Die Klonierung eines Gens
wiederum ist die Voraussetzung für dessen weitere Analyse, denn
durch Klonieren kann eine DNA in reiner Form gewonnen und vermehrt
werden.
Einzelne Schritte der Genklonierung im Ueberblick:
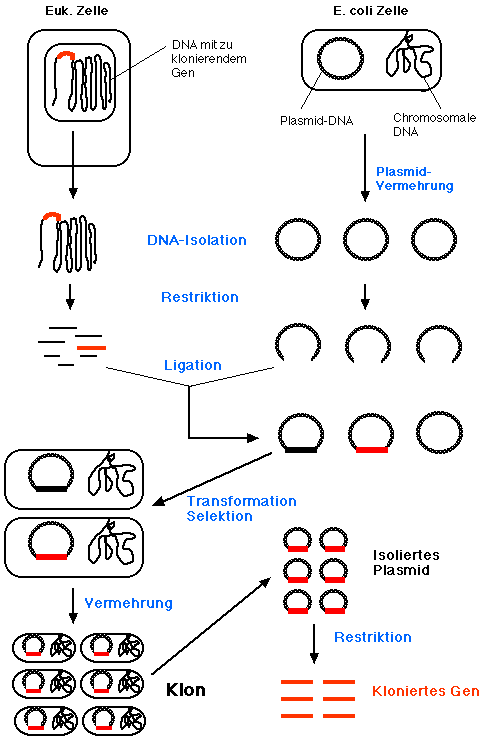
- Isolation von DNA (genomisch oder cDNA) und Plasmid-DNA
- Schneiden der DNA und des Plasmids mit einem Restriktionsenzym
- Ligation von DNA-Fragmenten mit der Plasmid-DNA
- Transformation von Bakterien mit rekombinantem Plasmid
- Selektion von Bakterienklonen, welche ein Plasmid aufgenommen
haben
- Isolierung des Klons mit dem gesuchten DNA-Fragment
DNA Klonierung: Warum?
Klonierung von genomischer DNA
Die totale DNA von Zellen der selben Art wird in Stücke
geschnitten und jedes Stück kloniert. Es resultiert eine
Sammlung von DNA-Fragmenten, welche das ganze Genom dieser Zellen
repräsentieren: die genomische DNA-Bank. Sie eignet sich zur
Isolation von Genen (mit Promoter, Introns etc.) und intergenischen
DNA-Sequenzen.
Klonierung von cDNA
Die totale mRNA von Zellen der selben Art wird isoliert, in cDNA
umgeschrieben und jede cDNA kloniert. Es resultiert eine Sammlung von
cDNA-Fragmenten, welche die gesamte mRNA dieser Zellen
repräsentieren: die cDNA-Bank. Sie eignet sich zur Isolation von
DNA-Abschnitten, welche für Eiweisse kodieren. Da die
kodierenden Sequenzen eines Genoms in der Regel nur wenige Prozente
der gesamten DNA einer Zelle darstellen, sind cDNA-Banken viel
kleiner (weniger Klone) als genomische Banken und eine
gewünschte (für Eiweiss kodierende) DNA-Sequenz kann daher
mit weniger Aufwand gefunden werden.
 top
top
DNA-Isolierung
Genomische DNA
Die wichtigsten Schritte:
- Zellen aufbrechen. Physikalisch: z.B. zerstossen der
gefrorenen Zellen im Mörser. Chemisch: z.B. Zell-Lyse mit
Detergenzien
- Abzentrifugieren von unlöslichen Zelltrümmern
- Reinigen der DNA. Abbau von Proteinen mit Proteasen.
Denaturieren und Ausfällen der Proteine mit Phenol. Die
Nucleinsäuren bleiben in Lösung. Auftrennen von
unlöslicher und löslicher Fraktion durch Zentrifugation.
Ribonuclease-Behandlung zum Abbau von RNA.
- Anreichern der DNA mittels Ethanol- oder
Isopropanolpräzipitation
Details : Chemie der
Nukleinsäuren
cDNA
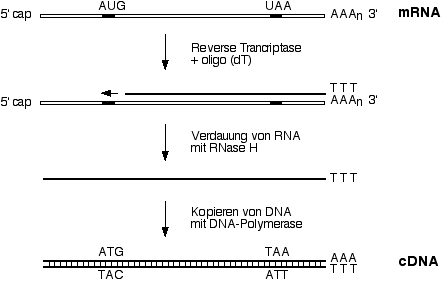
cDNA (komplementäre DNA) ist ein "Abdruck" der mRNA und
entspricht der DNA, welche für eine gesuchte mRNA codiert.
Die wichtigsten Schritte:
- Isolation aller RNAs einer Zelle, in der das gesuchte Gen
exprimiert, d.h. das Protein synthetisiert wird.
- Reinigung von mRNA (d.h. Abtrennen von tRNA und rRNA) durch
Affinitätschromatographie
- Synthese eines komplementären DNA-Stranges auf der
mRNA-Matrize: -> RNA-DNA-Doppelstrang
- Abbau des RNA-Stranges
- Synthese eines komplementären DNA-Stranges auf der
DNA-Matrize: -> cDNA (DNA-Doppelstrang)
Vorgehen:
- Isolierung der gesamten zellulären RNA (80% rRNA, 15%
tRNA, 5% mRNA)
- Abtrennen einer Fraktion, die hauptsächlich mRNA
enthält. Praktisch jedes mRNA-Molekül hat an seinem
3'-Ende eine Abfolge von Adenin-Nucleotiden = Poly (A)-Schwanz.
Dieser eröffnet einen sehr bequemen Weg, mRNA von der
restlichen Zell-RNA zu trennen. Man bindet
Desoxythymidin-Oligonucleotide ("Oligo(dT)") an Zellulose und
füllt diese in eine Säule. Lässt man die gesamte
Zell-RNA durch diese Säule laufen, bleiben die mRNAs mit
ihren Poly (A)-Schwänzen an den Oligonucleotiden
"hängen" (Bildung von A-T-Basenpaaren), während die
übrigen RNAs die Säule passieren. Nun lässt man
einen Puffer niedriger Ionenstärke durch die Säule
laufen. Dadurch lösen sich die Poly(A)-Oligo(dT)-Hybride auf,
und die gereinigte mRNA fliesst aus der Säule ab.
- cDNA-Synthese. Man gibt kurze Desoxythymidin-Oligonucleotide
zu der gereinigten mRNA. Diese hybridisieren mit den Poly
(A)-Schwänzen und wirken dann als Primer für die Reverse
Transcriptase, ein Enzym aus Retroviren. Mit Hilfe dieses Enzyms
werden mRNAs über RNA-DNA-Doppel-stränge zuerst in
einzelsträngige DNAs und darauf in doppelsträngige DNAs
überschrieben. cDNAs enthalten, wie Gene, die Information zur
Bildung von Proteinen, haben aber im Gegensatz zu Genen keine
Introns.
Detailiertere
Informationen
 top
top
Plasmid-Isolierung
Plasmide sind sich autonom vermehrende Minichromosomen. Sie werden
als Vektoren beim Klonieren benutzt.
Vorgehen:
- Plasmidhaltige Bakterien in einem Flüssig-Medium
züchten.
- Bakterien mit Lysozym behandeln, um die Zellwand zu
destabilisieren.
- Bakterien mit Detergens lysieren und im alkalischen pH
bakterielle DNA und Proteine denaturieren.
- Das Homogenat neutralisieren. Dabei fallen Proteine und
bakterielle genomische DNA aus und die Plasmide bleiben in
Lösung.
- Durch Zentrifugation Plasmide vom ausgefällten Material
trennen.
- Die Plasmide mit organischen Lösungsmitteln extrahieren
und mit Alkohol ausfällen.
|
|
Warum wird die bakterielle genomische DNA denaturiert
und die Plasmide bleiben in Lösung?
|
Lösung
|
Im Atelier gibt es ein Modell, an welchem diese
Vorgänge veranschaulicht werden.
|
 top
top